Comment visualiser les grappes dans un k-means modèle de l'apprentissage non supervisé
L'ensemble de données Iris est pas facile à tracer pour l'analyse prédictive dans sa forme originale. Par conséquent, vous devez réduire le nombre de dimensions en appliquant une algorithme de réduction de dimensionnalité qui fonctionne sur tous les quatre numéros et deux sorties de nouveaux numéros (qui représentent les quatre numéros originaux) que vous pouvez utiliser pour faire de la parcelle.
| Longueur des sépales | Sépale Largeur | Pétale Longueur | Pétale Largeur | Classe cible / Étiquette |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0,2 | Setosa (0) |
| 7.0 | 3.2 | 4.7 | 1.4 | Versicolor (1) |
| 6.3 | 3.3 | 6.0 | 2.5 | Virginica (2) |
Le code suivant fera la réduction de dimension:
>>> From sklearn.decomposition importation PCA >>> from sklearn.datasets importer load_iris >>> iris = load_iris () >>> pca = PCA (n_components = 2) .fit (iris.data) >>> pca_2d = pca .transform (iris.data)
Lignes 2 et 3 charge l'ensemble de données Iris.
Après vous exécutez le code, vous pouvez taper le pca_2d variable dans l'interprète et il sera tableaux de sortie (penser à une tableau comme un conteneur d'éléments dans une liste) avec deux points au lieu de quatre. Maintenant que vous avez l'ensemble des fonctionnalités réduites, vous pouvez tracer les résultats avec le code suivant:
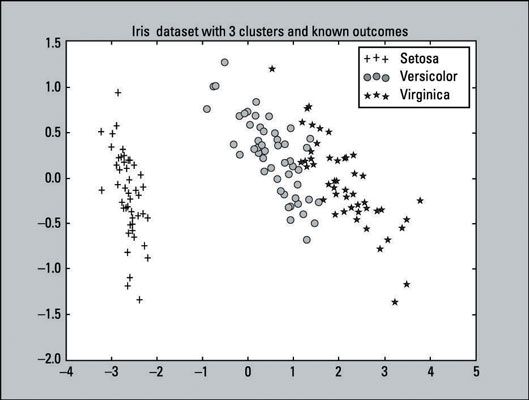
>>> Pylab d'importation comme pl >>> for i in range (0, pca_2d.shape [0]): >>> si iris.target [i] == 0: >>> c1 = pl.scatter (pca_2d [ i, 0], pca_2d [i, 1], c = 'r', marqueur = '+') >>> Elif iris.target [i] == 1: >>> c2 = pl.scatter (pca_2d [i , 0], pca_2d [i, 1], c = 'g', marqueur = 'o') >>> Elif iris.target [i] == 2: >>> c3 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c = 'b', marqueur = '*') >>> pl.legend ([C1, C2, C3], ['Setosa »,« versicolor', 'Virginica'] ) >>> pl.title ('ensemble de données Iris avec 3 clusters et knownoutcomes') >>> pl.show ()La sortie de ce code est un terrain qui devrait être similaire au graphique ci-dessous. Ceci est un tracé représentant comment les résultats connus de l'ensemble de données Iris devraient ressembler. Il est ce que vous aimeriez le regroupement K-means à atteindre.
L'image montre un nuage de points, ce qui est un graphique de points tracés représentant une observation sur un graphique, de tous les 150 observations. Comme indiqué sur le graphique représente et la légende:
Il ya 50 points positifs qui représentent la Setosa classe.
Il ya 50 cercles qui représentent la Classe versicolor.
Il ya 50 étoiles qui représentent la Classe Virginica.
Le graphique ci-dessous montre une représentation visuelle des données que vous demandez K-means à se regrouper: un nuage de points avec 150 points de données qui ont pas été étiquetés (d'où tous les points de données sont de la même couleur et de la forme). Le K-means ne sais pas quelle cible Résultats- les données réelles que nous sommes en cours d'exécution à travers l'algorithme n'a pas eu sa dimension encore réduit.
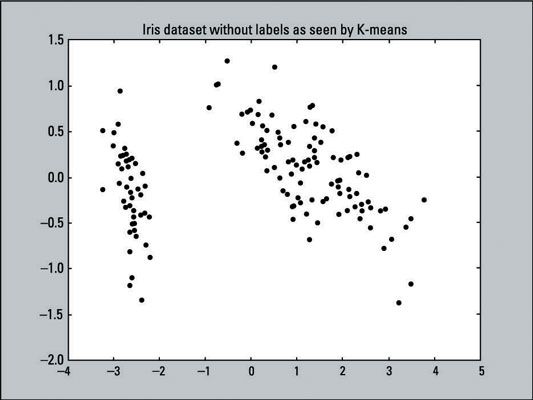
La ligne de code suivante crée ce diagramme de dispersion, en utilisant les valeurs X et Y de pca_2d et la coloration tous les points de données en noir (c = 'black' définit la couleur au noir).
>>> Pl.scatter (pca_2d [:, 0], pca_2d [:, 1], c = 'noir') >>> pl.show ()
Si vous essayez ajustant les données en deux dimensions, qui a été réduit par l'APC, l'algorithme k-means échouera à se regrouper correctement les classes Virginica et Versicolor. Utilisation de PCA pour prétraiter les données vont détruire trop d'informations que K-means besoins.
Après K-means a équipé les données Iris, vous pouvez faire un diagramme de dispersion des grappes que l'algorithme produced- suffit d'exécuter le code suivant:
>>> For i in range (0, pca_2d.shape [0]): >>> si kmeans.labels_ [i] == 1: >>> c1 = pl.scatter (pca_2d [i, 0], pca_2d [ i, 1], c = 'r', marqueur = '+') >>> Elif kmeans.labels_ [i] == 0: >>> c2 = pl.scatter (pca_2d [i, 0], pca_2d [i , 1], c = 'g', marqueur = 'o') >>> Elif kmeans.labels_ [i] == 2: >>> c3 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c = 'b', marqueur = '*') >>> pl.legend ([C1, C2, C3], ['Cluster 1 »,« Cluster 0', 'Cluster 2']) >>> pl.title («K-means grappes dans l'ensemble de données Iris 3clusters ') >>> pl.show ()
Rappelons que K-means étiquetés les 50 premières observations avec l'étiquette de 1, la deuxième étiquette de 50 à 0, et le dernier 50 avec l'étiquette de 2. Dans le code, simplement donné, les lignes avec le si, Elif, et légende Comptes (lignes 2, 5, 8, 11) reflète ces étiquettes. Ce changement a été fait pour le rendre facile de comparer avec les résultats réels.
La sortie du diagramme de dispersion est montré ici:
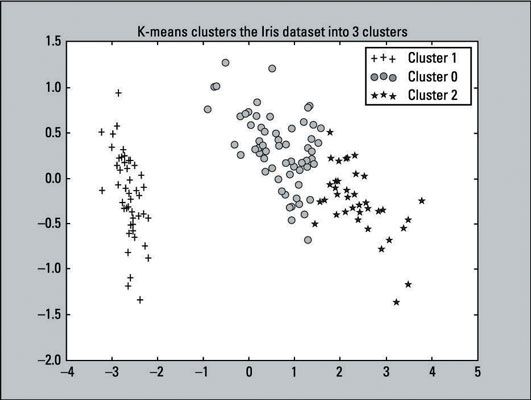
Comparez les k-moyennes sortie vers le nuage de points initial - qui fournit les étiquettes parce que les résultats sont connus. Vous pouvez voir que les deux parcelles se ressemblent. L'algorithme K-means a fait un très bon travail avec le clustering. Bien que les prévisions ne sont pas parfaits, ils se rapprochent. Voilà une victoire pour l'algorithme.
Dans l'apprentissage non supervisé, vous obtenez rarement une sortie qui est 100 pour cent exact, car les données du monde réel est rarement aussi simple que cela. Vous ne saurez pas combien de grappes de choisir (ou de tout paramètre d'initialisation pour d'autres algorithmes de clustering). Vous aurez à gérer les valeurs aberrantes (points de données qui ne semblent pas compatibles avec les autres) et des ensembles de données complexes qui sont denses et non linéairement séparables.
Vous ne pouvez arriver à ce point si vous savez combien de grappes l'ensemble de données a. Vous ne devez pas vous inquiéter sur les fonctionnalités à utiliser ou à réduire les dimensions d'un ensemble de données qui a si peu de caractéristiques (dans ce cas, quatre). Cet exemple seulement réduit les dimensions pour le bien de visualiser les données sur un graphique. Il ne correspondait pas au modèle avec l'ensemble de données de dimension réduite.
Voici la liste complète du code qui crée les deux diagrammes de dispersion et les codes-couleurs des points de données:
>>> From sklearn.decomposition importation PCA >>> from KMeans d'importation sklearn.cluster >>> from sklearn.datasets importer load_iris >>> pylab d'importation comme pl >>> iris = load_iris () >>> pca = PCA (n_components = 2) .fit (iris.data) >>> pca_2d = pca.transform (iris.data) >>> pl.figure («Référence Plot ') >>> pl.scatter (pca_2d [:, 0], pca_2d [:, 1], c = iris.target) >>> kmeans = KMeans (n_clusters = 3, random_state = 111) >>> kmeans.fit (iris.data) >>> pl.figure («K-means avec 3 clusters ») >>> pl.scatter (pca_2d [:, 0], pca_2d [:, 1], c = kmeans.labels _) >>> pl.show ()



