Comment visualiser le classificateur dans un modèle d'apprentissage supervisé svm
L'ensemble de données Iris est pas facile à tracer pour l'analyse prédictive dans sa forme originale, parce que vous ne pouvez pas tracer les quatre coordonnées (des fonctions) de l'ensemble de données sur un écran en deux dimensions. Par conséquent, vous devez réduire les dimensions en appliquant une la réduction de la dimensionnalité algorithme pour les caractéristiques.
Dans ce cas, l'algorithme que vous allez utiliser pour faire la transformation de données (en réduisant les dimensions des caractéristiques) est appelée Analyse en Composantes Principales (ACP).
| Longueur des sépales | Sépale Largeur | Pétale Longueur | Pétale Largeur | Classe cible / Étiquette |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0,2 | Setosa (0) |
| 7.0 | 3.2 | 4.7 | 1.4 | Versicolor (1) |
| 6.3 | 3.3 | 6.0 | 2.5 | Virginica (2) |
L'algorithme PCA prend toutes les quatre caractéristiques (nombres), fait un peu de maths sur eux, et sort deux nouveaux numéros que vous pouvez utiliser pour faire de la parcelle. Pensez PCA comme suivant deux étapes générales:
Il prend en entrée un ensemble de données avec de nombreuses fonctionnalités.
Il réduit cette entrée à un plus petit ensemble de fonctions en transformant les composants de la fonctionnalité mis en ce qu'il considère comme les principaux (principaux) des composants (ou un algorithme déterminé défini par l'utilisateur).
Cette transformation de l'ensemble des fonctionnalités est également appelé extraction de caractéristiques. Le code suivant fait la réduction de dimension:
>>> From sklearn.decomposition importation PCA >>> pca = PCA (n_components = 2) .fit (X_train) >>> pca_2d = pca.transform (X_train)
Si vous avez déjà importé des bibliothèques ou des ensembles de données, il est pas nécessaire de ré-importer ou les charger dans votre session actuelle de Python. Si vous le faites, cependant, il ne devrait pas affecter votre programme.
Après vous exécutez le code, vous pouvez taper le pca_2d variable dans l'interprète et de voir qu'il émet des tableaux avec deux points au lieu de quatre. Ces deux nouveaux numéros sont des représentations mathématiques des quatre anciens numéros. Lorsque la fonction de réduction défini, vous pouvez tracer les résultats en utilisant le code suivant:
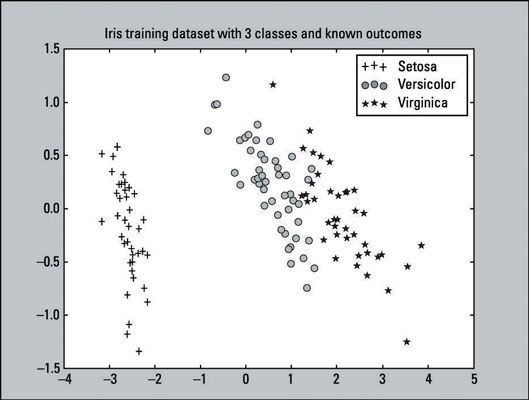
>>> Pylab d'importation comme pl >>> for i in range (0, pca_2d.shape [0]): >>> si y_train [i] == 0: >>> c1 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c = 'r', marqueur = '+') >>> Elif y_train [i] == 1: >>> c2 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c = 'g', marqueur = 'o') >>> Elif y_train [i] == 2: >>> c3 = pl.scatter (pca_2d [i, 0], pca_2d [i , 1], c = 'b', marqueur = '*') >>> pl.legend ([C1, C2, C3], ['Setosa »,« versicolor', 'Virginica']) >>> pl. titre ('ensemble de données de formation Iris avec 3 classes andknown résultats ») >>> pl.show ()C'est un nuage de points - une visualisation des points tracés représentant observations sur un graphique. Ce nuage de points représente notamment les résultats connus de l'ensemble de données de formation Iris. Il ya 135 points tracés (observations) de notre ensemble de données de formation. L'ensemble de données de formation se compose de
45 avantages que représentent la classe Setosa.
48 cercles qui représentent la classe versicolor.
42 étoiles qui représentent la classe Virginica.
Vous pouvez confirmer le nombre indiqué de classes en entrant le code suivant:
>>> Somme (y_train == 0) 45 >>> somme (y_train == 1) 48 >>> somme (y_train == 2) 42
De cette parcelle, vous pouvez clairement dire que la classe Setosa est linéairement séparable des deux autres classes. Alors que les classes Versicolor et Virginica ne sont pas complètement séparés par une ligne droite, ils ne sont pas de chevauchement par beaucoup. Du point de vue visuel simple, les classificateurs devraient faire assez bien.
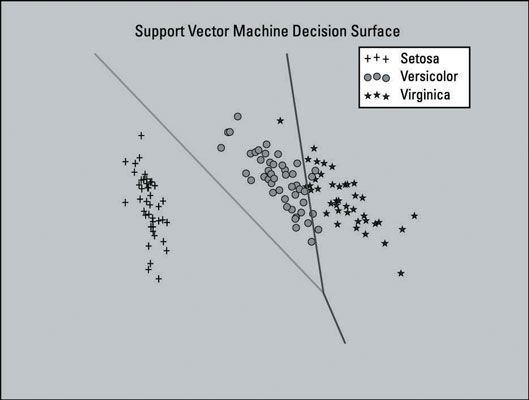
L'image ci-dessous montre un graphique de la (SVM) modèle Support Vector Machine formé avec un ensemble de données qui a été réduite à deux dimensions caractéristiques. Quatre caractéristiques est une petite fonction réglage dans ce cas, vous voulez garder tous les quatre de sorte que les données peuvent conserver la plupart de ses informations utiles. L'intrigue est montré ici comme aide visuelle.
Cette parcelle comprend le surface de décision pour le classificateur - la zone dans le graphique qui représente la fonction de décision que SVM utilise pour déterminer le résultat de la nouvelle entrée de données. Les lignes séparent les zones où le modèle permettra de prédire la classe particulière qui d'un point de données appartient.
La partie gauche de la parcelle permettront de prédire la classe Setosa, la section du milieu permettra de prédire la classe versicolor, et la section de droite permettra de prédire la classe Virginica.
Le modèle SVM que vous avez créé ne pas utiliser l'ensemble des fonctionnalités de dimensions réduites. Ce modèle utilise uniquement la réduction de la dimensionnalité ici pour générer un tracé de la surface de décision du modèle SVM - comme aide visuelle.
La liste complète du code qui crée l'intrigue est fourni comme référence. Il ne devrait pas être exécuté en séquence avec notre exemple, si vous suivez. Il peut remplacer certaines des variables que vous pouvez déjà avoir dans la session.
Le code pour produire ce complot est basé sur l'exemple de code fourni sur le scikit apprendre site Internet. Vous pouvez en apprendre plus sur la création des parcelles comme celles-ci à la scikit-learn site Internet.
Voici la liste complète du code qui crée l'intrigue:
>>> De PCA sklearn.decomposition d'importation de sklearn.datasets >>> >>> importer load_iris de sklearn importation svm >>> from sklearn importation cross_validation >>> pylab d'importation comme pl >>> numpy d'importation comme np >>> iris = load_iris () >>> X_train, X_test, y_train, y_test = cross_validation.train_test_split (iris.data, iris.target, test_size = 0,10, random_state = 111) >>> pca = PCA (n_components = 2) .fit (X_train ) >>> pca_2d = pca.transform (X_train) >>> svmClassifier_2d = svm.LinearSVC (random_state = 111) .fit (pca_2d, y_train) >>> for i in range (0, pca_2d.shape [0]): >>> si y_train [i] == 0: >>> c1 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c = 'r', s = 50, marqueur = '+' ) >>> Elif y_train [i] == 1: >>> c2 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c = 'g', S = 50, marqueur = 'o ') >>> Elif y_train [i] == 2: >>> c3 = pl.scatter (pca_2d [i, 0], pca_2d [i, 1], c =' b ', S = 50, marqueur =' * ') >>> pl.legend ([c1, c2, c3], [' Setosa ',' Versicolor ',' Virginica ']) >>> x_min, x_max pca_2d = [0 :,] .min () - 1, pca_2d [:, 0] .max () + 1 >>> Y_MIN, y_max = pca_2d [:, 1] .min () - 1, pca_2d [:, 1] .max () + 1 >>> xx , aa = np.meshgrid (np.arange (x_min, x_max, 0,01), np.arange (Y_MIN, y_max, 0,01)) >>> Z = svmClassifier_2d.predict (np.c_ [xx.ravel (), yy.ravel ()]) >>> Z = Z.reshape (xx.shape) >>> pl.contour (xx, yy, Z) >>> pl.title («Support Vector Machine décision Surface ') >> > pl.axis («off») >>> pl.show ()




